我校园艺园林学院王傲雪教授团队在Horticulture Research(IF=7.6,中科院1区Top期刊)上线了题为“The nature of complex structural variations in tomatoes”的研究论文,研究以模式植物番茄作为对象,深入分析了SV及其上下游序列特征,提出了新的识别策略,系统解决了复杂SV在重复区域中的鉴定难题,并为植物组学研究提供了一个高精度的SV基准数据集,有效弥补了作物改良中基因组基础薄弱的短板。

结构变异(Structural variations,SVs)作为基因组中大片段的遗传变异,能够通过改变调控元件和三维结构来扰乱基因的表达和功能。随着基因组学在育种领域的迅猛发展,SVs对作物重要农艺性状的作用尤为关键。然而,相比于单核苷酸多态性等小变异,全基因组范围的SVs精准鉴定一直面临挑战,尤其是在重复序列丰富的区域,复杂SV区域和分类的界定尤为困难。现有基准数据集(Benchmark)多排除了这些区域,导致检测准确率被严重高估,亟需更加全面、标准的SV参考。
团队基于两套高质量番茄基因组,整合了14种主流SV检测工具的结果,研究总结出鉴定误差主要的三大来源为:边界定位不准、断点分辨率不足、类型定义不一致。团队提出优先识别“唯一比对序列”(Uniquely Aligned Sequences, UAS)的人工检验方法,最终构建了包含1635个碱基级别SVs的Benchmark。基于此,研究人员提出了结构变异的三个特征:一是根据SVs周围丰富的拷贝片段变化,将断点分类成四种模式;二是定义了插入、缺失、倒位和替换为SV的基本类型,有望简化图泛基因组的构建;三是SV偏好发生于富含AT重复的基因调控区域内,且与环境适应性相关。
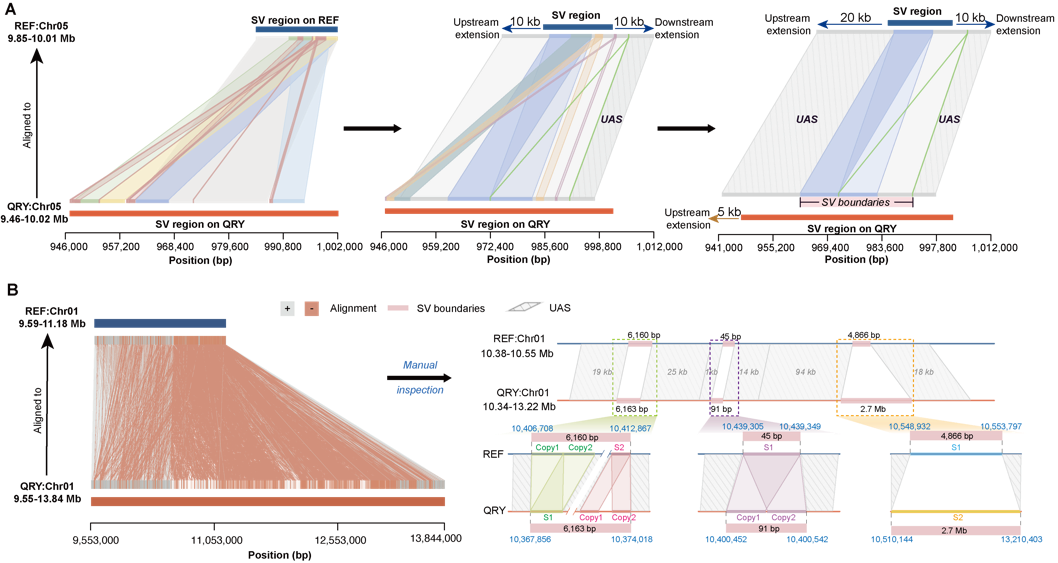
利用新的Benchmark重新评估现有检测算法,最佳的F1-score仅为53.77%,凸显了对高度精确检测算法的迫切需求。此外,利用人工检验流程,在25个番茄样本中成功鉴定出与TomLoxC基因相关的16个新的SVs,为今后番茄风味的研究奠定基础。
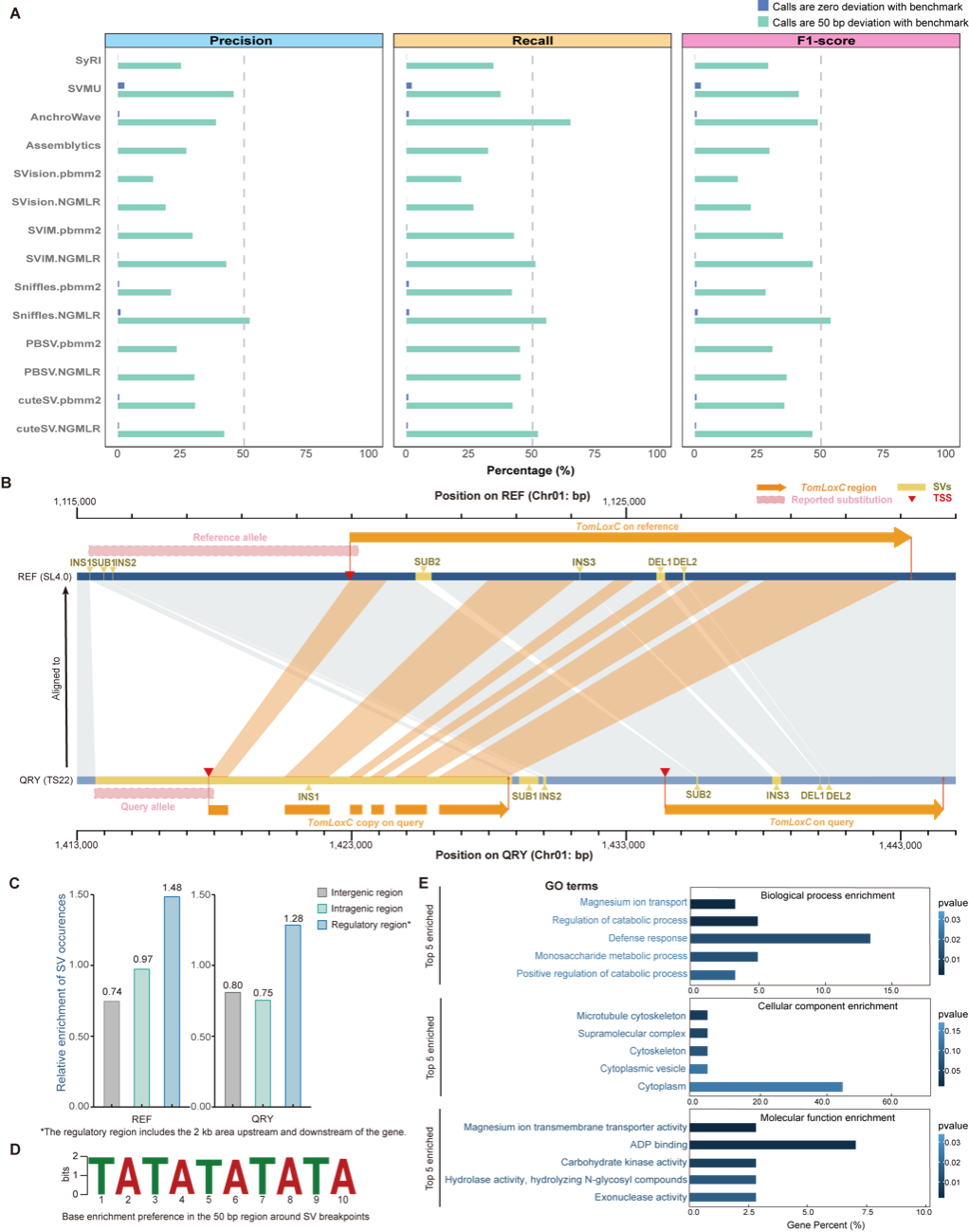
总体而言,该研究成果有助于提高对SV的认识,为开发下一代SV检测算法和推进基因组应用提供了有力参考,有助于未来作物的基因组育种工作。
我校园艺园林学院博士生崔雪、刘宇欣,硕士生赵启悦以及中国科学院植物研究所博士生孙淼为文章共同第一作者。东北农业大学园艺园林学院王傲雪教授、Hossein Ghanizadeh教授和中国科学院植物研究所周姚研究员为共同通讯作者。该研究得到了国家自然科学基金、黑龙江省农业农村厅智能分子育种项目、黄三角国家农高区省级科技创新发展专项资金和中科院战略性先导科技专项的支持。
王傲雪简介:二级教授,博士生导师,黑龙江省高层次人才(B类),国家政府特殊津贴获得者。主持国家重点研发计划、国家自然基金等26项科研项目,获得省部级以上奖励9项。获得发明专利21项,农业农村部生物肥料证书3个,出版专著及教材10部,发表SCI论文70余篇。
(供稿/园艺园林学院)